หน่วยรับกลิ่น
หน่วยรับกลิ่น หรือ ตัวรับกลิ่น (อังกฤษ: Olfactory receptor ตัวย่อ OR) เป็นโปรตีนหรือหน่วยรับความรู้สึกซึ่งแสดงออกที่เยื่อหุ้มเซลล์ของเซลล์ประสาทรับกลิ่น และมีหน้าที่ตรวจจับกลิ่น (คือสารประกอบที่มีกลิ่น) ซึ่งทำให้รู้กลิ่นได้ การทำงานของหน่วยรับกลิ่นจะจุดชนวนการถ่ายโอนกลิ่นเป็นกระแสประสาทภายในเซลล์ซึ่งส่งข้อมูลกลิ่นไปยังสมอง หน่วยรับกลิ่นเป็นสมาชิกที่คล้ายกับ rhodopsin ในชั้น A (class A rhodopsin-like) ของกลุ่มโปรตีน G protein-coupled receptor (GPCR)[2][3] โดยมีลำดับโปรตีนต่างหากภายในกลุ่มที่ไม่เหมือนกับสมาชิกของ GPCR อื่น ๆ[4] หน่วยรับกลิ่นเป็นหมู่ยีน (multigene) ที่มียีนถึง 800 ยีนในมนุษย์ และ 1,400 ในหนูหริ่ง[5] แต่ยีนเหล่านี้ไม่ได้เข้ารหัสโปรตีนคือไม่ได้ใช้งานได้ทุกยีน หนูหริ่งมียีน OR ที่เข้ารหัสโปรตีนถึง 1,035 ยีน เทียบกับมนุษย์ที่มีเพียงแค่ 387 ยีน[6]
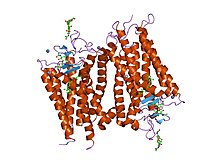

การแสดงออก
แก้ในสัตว์มีกระดูกสันหลัง หน่วยรับกลิ่นจะอยู่ที่ทั้งซีเลียและไซแนปส์ของเซลล์ประสาทรับกลิ่น[7] โดยเซลล์รับกลิ่นหนึ่ง ๆ จะแสดงออกหน่วยรับกลิ่นประเภทเดียวเท่านั้น[8] และจะอยู่ในเยื่อบุผิวของทางเดินอากาศในมนุษย์ด้วย[9]
ในแมลง หน่วยรับกลิ่นจะอยู่ที่หนวดและอวัยวะรับสารเคมีอื่น ๆ[10] ตัวอสุจิยังแสดงออกหน่วยรับกลิ่นอีกด้วย ซึ่งเชื่อว่ามีบทบาทในการเคลื่อนที่ตอบสนองต่อสารเคมี (chemotaxis) เพื่อตรวจหาเซลล์ไข่[11]
ในหนูหริ่ง
แก้มีการศึกษาการวางระเบียบของหน่วยรับกลิ่นในหนูหริ่ง คือ ในหนูหริ่ง หน่วยรับกลิ่นหนึ่ง ๆ จะแสดงออกที่เยื่อรับกลิ่นที่แบ่งเป็นโซนคร่าว ๆ หลายโซน หน่วยแต่ละอย่างจะแสดงออกที่เซลล์รับกลิ่นประมาณ 5,000 ตัวที่กระจายไปโดยสุ่มภายในโซน ดังนั้น เซลล์ที่มีหน่วยรับกลิ่นต่างกันจะอยู่อย่างผสมผเสกัน โซนหนึ่ง ๆ อาจจะมีหน่วยรับกลิ่นหนึ่ง ๆ มากกว่าโซนอื่น ๆ แต่โซนหนึ่ง ๆ ก็จะมีหน่วยรับกลิ่นที่แสดงออกหลายประเภท แม้ว่าการแบ่งโซนเช่นนี้จะยังไม่ชัดเจนว่ามีเหตุทางวิวัฒนาการอย่างไร แต่เพราะโซนหนึ่ง ๆ จะส่งแอกซอนไปยังส่วนโดยเฉพาะ ๆ ของป่องรับกลิ่น นักวิชาการจึงเชื่อว่า นี่เป็นการวางรูปแบบโดยเฉพาะ ๆ ที่มีผลต่อการประมวลข้อมูลกลิ่น นอกจากนั้น การมีหน่วยรับกลิ่นในหลาย ๆ โซนก็มีผลว่า ความเสียหายต่อโซนหนึ่งจะไม่ทำลายการได้กลิ่นหนึ่ง ๆ โดยสิ้นเชิงเพราะยังมีหน่วยรับกลิ่นนั้น ๆ เหลืออยู่ในโซนอื่น ๆ[12]
โครงสร้าง
แก้เหมือนกับ G protein-coupled receptor (GPCR) อื่น ๆ หน่วยรับกลิ่นมีบริเวณไม่ชอบน้ำ 7 บริเวณที่น่าจะใช้เป็นโดเมนข้ามเยื่อหุ้มเซลล์ (transmembrane domain) ซึ่งมีรูปเกลียว (helix) งานศึกษา GPCR แสดงนัยว่า การผสมรวมกันของโดเมนข้ามเยื่อหุ้มเซลล์จะสร้าง "กระเป๋า" ที่เป็นจุดยึดกับโมเลกุลกลิ่น และเนื่องจากลำดับกรดอะมิโนของโดเมนข้ามเยื่อหุ้มเซลล์หลายตัวมีความหลากหลายมาก นักวิชาการจึงเชื่อว่า นี่อาจเป็นเป็นมูลฐานให้เกิดความหลากหลายของกระเป๋าที่สามารถจับกับลิแกนด์กลิ่นต่าง ๆ ได้[4]
ความรู้เรื่องลำดับโปรตีนหลัก ๆ ของหน่วยรับกลิ่นเป็นพัน ๆ ได้มาจากการศึกษาจีโนมของสิ่งมีชีวิตมากกว่าโหล ถึงแม้โดยเดือนพฤษภาคม ค.ศ. 2016 ก็ยังไม่มีการแสดงโครงสร้างของ OR ให้ชัดเจน เหมือนกับ GPCR ๆ อื่น ยังไม่มีการแสดงโครงสร้างของหน่วยรับกลิ่นในระดับอะตอม และข้อมูลเกี่ยวกับโครงสร้างทั้งหมดก็มาจากแบบจำลองโดย homology modeling[13] คือลำดับโปรตีนของหน่วยรับกลิ่นจะเป็นเหมือนกับ class A GPCR ซึ่งอาจใช้จำลองโครงสร้างของหน่วยรับกลิ่นได้[14]
งานศึกษาปี 2015 แสดงว่า กลไกของการรู้จักลิแกนด์ แม้จะเหมือนกับ class A GPCR ที่ไม่ได้เป็นตัวรับกลิ่นอื่น ๆ แต่ก็ยังต้องอาศัยโปรตีนที่เฉพาะกับหน่วยรับกลิ่นเท่านั้น โดยเฉพาะในโดเมนรูปเกลียวที่หก[15]
มีลำดับโปรตีนที่เหมือนกันอย่างยิ่งใน 3/4 ของ OR ทั้งหมดที่เป็นจุดยึดไอออนโลหะแบบมีสามขา[16] นักวิชาการจึงเสนอสมมติฐานว่า OR จริง ๆ เป็น metalloprotein (น่าจะประกอบด้วยไออนสังกะสี ทองแดง และอาจจะแมงกานีส) ที่ทำหน้าที่เป็นจุดยึดโมเลกุลกลิ่นหลายอย่างโดยเป็น Lewis acid site งานปี ค.ศ. 1978 ได้เสนอว่า Cu (I) มีโอกาสเป็น metallo-receptor site ของระบบรู้กลิ่นมากที่สุด สำหรับสารระเหยกลิ่นแรงซึ่งเป็นลิแกนด์ที่สามารถจับกับโลหะโดย coordinate covalent bond ได้ดีด้วย (เช่น thiol)[17]
ต่อมางานศึกษาปี 2012 ก็ได้ให้หลักฐานกับสมมติฐานนี้สำหรับ OR ของหนูในกรณีเฉพาะ คือ MOR244-3 โดยแสดงว่า ทองแดงเป็นส่วนประกอบสำคัญในการตรวจจับ thiol และสารประกอบที่มีกำมะถันบางอย่าง คือเมื่อใช้สารเคมีจับกับทองแดงในจมูกหนูเพื่อไม่ให้หน่วยรับความรู้สึกมีใช้ ผู้ทำงานวิจัยก็ได้พบว่า หนูไม่สามารถตรวจจับ thiole ได้ แต่ผู้ทำงานวิจัยนี้ก็พบด้วยว่า MOR244-3 ไม่มีจุดยืดไอออนโลหะโดยเฉพาะดังที่เสนอโดยสมมติฐาน แต่กลับมีลักษณะที่ต่างกันใน EC2 domain[18]
กลไก
แก้แทนที่จะเข้ายึดกับลิแกนด์โดยเฉพาะ ๆ หน่วยรับกลิ่นแต่ละอย่างจะมีสัมพรรคภาพ (affinity) กับโมเลกุลกลิ่นต่าง ๆ ในระดับต่าง ๆ กัน ดังนั้น ในนัยกลับกัน โมเลกุลกลิ่นเดียวอาจจะยึดกับหน่วยรับกลิ่นจำนวนหนึ่งโดยมีสัมพรรคภาพในระดับต่าง ๆ กัน[19] ซึ่งขึ้นอยู่กับคุณสมบัติทางกายภาพ-เคมีเช่นปริมาตรของโมเลกุล[20]
หน่วยรับกลิ่นที่จับกับโมเลกุลกลิ่นจะทำให้เกิดการส่งสัญญาณเป็นลำดับภายในเซลล์ ซึ่งในที่สุดก็ทำให้เซลล์ลดขั้วแล้วส่งศักยะงานไปยังป่องรับกลิ่น[4] โดยรายละเอียดก็คือ เมื่อจับกับกลิ่นแล้ว หน่วยรับกลิ่นจะเปลี่ยนโครงสร้างแล้วเริ่มการทำงานของ G protein ภายในเซลล์รับกลิ่นซึ่งอยู่ที่ปลาย carboxyl ของหน่วยรับกลิ่น G protein (Golf และ/หรือ Gs)[21] ซึ่งเป็นประเภทที่เฉพาะต่อระบบรับกลิ่น ก็จะเริ่มการทำงานของเอนไซม์ adenylate cyclase III (ACIII) ซึ่งเป็นเอนไซม์เฉพาะในระบบรับกลิ่นเช่นกัน และเพิ่มการปล่อย cyclic AMP (cAMP) ซึ่งทำหน้าที่เป็น second messenger โดยอาศัยอะดีโนซีนไตรฟอสเฟต (ATP) cAMP ก็จะเปิดช่องไอออน cyclic nucleotide-gated ion channel ทำให้ไอออนแคลเซียม (Na+) และโซเดียม (Ca2+) ซึมเข้ามาในเซลล์ได้ แล้วทำให้เซลล์รับกลิ่นลดขั้ว (depolarized) นอกจากนั้น Ca2+ ที่เพิ่มขึ้นก็จะเปิดช่องไอออน Ca2+-gated Cl- channel ซึ่งขยายการลดขั้วของเซลล์ที่แพร่กระจายไปตามตัวเซลล์อย่างแพสซิฟจนถึงส่วน axon hillock ของตัวเซลล์ เป็นจุดที่สร้างศักยะงานอาศัยช่องไอออน voltage-regulated Na+ channel เพื่อส่งไปยังป่องรับกลิ่น[22]
ทั้งโปรตีน Golf และเอนไซม์ ACIII จะมีอยู่ที่ซีเลียและปุ่มเดนไดรต์ของเซลล์รับกลิ่นเท่านั้น ซึ่งเป็นการยืนยันว่าการรับรู้กลิ่นจะเริ่มที่ซีเลียของเซลล์รับกลิ่นเป็นจุดเบื้องต้น[22]
ในงานที่ตีความข้อมูลแบบถกเถียงกันไม่จบปี 2007 ผู้เขียนคาดว่า หน่วยรับกลิ่นจริง ๆ อาจจะรับรู้พลังงานสั่นในระดับต่าง ๆ ของโมเลกุลผ่านกลไก quantum coherence แทนที่จะรับรู้ลักษณะทางโครงสร้างของโมเลกุล[23] โดยมีหลักฐานแสดงว่า แมลงวันสามารถแยกแยะโมเลกุลกลิ่นสองอย่างที่ต่างกันโดยเพียงแค่ไอโซโทปไฮโดรเจน ซึ่งเป็นตัวทำให้ระดับพลังงานสั่นของโมเลกุลต่างกันมากด้วย[24] อนึ่ง แมลงวันไม่เพียงแยกแยะโมเลกุลกลิ่นหนึ่ง ๆ ที่มีดิวเทอเรียม (ไอโซโทปหนึ่งของไฮโดรเจน) หรือไม่มี แต่สามารถแยกแยะโมเลกุลกลิ่นใหม่ ๆ ที่มีดิวเทอเรียมหรือไม่มีได้ด้วย นอกจากนั้น แมลงวันยังสามารถเรียนรู้หลีกเลี่ยงโมเลกุลกลิ่นที่แม้ไม่มีดิวเทอเรียมแต่ก็ยังมีพลังงานสั่นบางส่วนเหมือนกับโมเลกุลที่มีดิวเทอเรียม นี่เป็นหลักฐานที่คำอธิบายทางโครงสร้างของการมีหรือไม่มีดิวเทอเรียมไม่สามารถอธิบายได้
แต่ก็ควรจะสังเกตว่า การเพิ่มดิวเทอเรียมยังเปลี่ยนความร้อนเนื่องจากการดูดซับ เปลี่ยนจุดเดือดและจุดหลอมเหลวของโมเลกุล (จุดเดือด - 100.0 °C สำหรับ H2O เทียบกับ 101.42 °C สำหรับ D2O; จุดหลอมเหลว - 0.0 °C สำหรับ H2O 3.82 °C สำหรับ D2O), เปลี่ยน pKa คือ ค่าคงที่การแตกตัวของกรด (ค่าคงที่การแตกตัว 9.71x10−15 สำหรับ H2O เทียบกับ 1.95x10−15 สำหรับ D2O) และเปลี่ยนกำลังพันธะของไฮโดรเจนอีกด้วย ปรากฏการณ์เนื่องจากไอโซโทปเช่นนี้สามัญมาก และก็รู้กันเป็นอย่างดีว่า การทดแทนด้วยดิวเทอเรียมก็จะเปลี่ยนค่าคงตัวทางการยึดเหนี่ยวระหว่างโมเลกุลกลิ่นกับโปรตีนรับกลิ่น[25]
มีการอ้างว่า หน่วยรับกลิ่นของมนุษย์สามารถแยกแยะ isotopomer ที่มีหรือไม่มีดิวเทอเรียมของ cyclopentadecanone โดยระดับพลังการสั่น[26] แต่ก็มีรายงานขัดแย้งว่า OR5AN1 ที่เป็นหน่วยรับกลิ่นในมนุษย์และตอบสนองอย่างมีกำลังต่อ cyclopentadecanone และ muscone กลับไม่สามารถแยกแยะ isotopomer ของสารประกอบเหล่านี้นอกกาย (in vitro) นอกจากนั้น ยังพบด้วยว่า หน่วยรับ methanethiol คือ MOR244-3 ของหนูหริ่ง บวกกับหน่วยรับกลิ่นอื่น ๆ ของมนุษย์และหนูหริ่ง จะตอบสนองคล้าย ๆ กันต่อ isotopomer ของลิแกนด์ธรรมดา ลิแกนด์ที่มีดิวเทอเรียม และลิแกนด์ที่มี carbon-13 ซึ่งเป็นผลคล้ายกับที่พบใน OR5AN1 ของหนู[27] ดังนั้น นักวิจัยจึงได้สรุปว่า ทฤษฎีแรงสั่นที่เสนอใช้ไม่ได้กับหน่วยรับกลิ่น OR5AN1 ของมนุษย์, หน่วยรับ thiol MOR244-3 ของหนู, หรือหน่วยรับกลิ่นอื่น ๆ ที่ได้ตรวจสอบ
นอกจากนั้น กลไกการถ่ายโอนอิเล็กตรอนเนื่องจากความถี่แรงสั่นของโมเลกุลกลิ่นตามที่เสนอ ก็ยังสามารถขัดขวางได้ง่าย ๆ โดยปรากฏการณ์ควอนตัมของแรงสั่นโมเลกุลที่ไม่ใช่กลิ่นอื่น ๆ ดังนั้น จึงมีหลักฐานหลายแนวที่ค้านทฤษฎีแรงสั่นเกี่ยวกับกลิ่น[28]
แต่งานศึกษาหลังก็ถูกวิจารณ์ว่า ใช้ "เซลล์ในหลอดทดลองแทนที่จะใช้สิ่งมีชีวิตทั้งตัว" และว่า "การแสดงออกของยีนหน่วยรับกลิ่นในเซลล์ไตมนุษย์ระยะตัวอ่อน ไม่ได้เลียนแบบธรมชาติที่ซับซ้อนของการได้กลิ่น" ผู้เขียนจึงกล่าวตอบโต้ว่า "เซลล์ไตระยะตัวอ่อนไม่ได้เหมือนกับเซลล์ในจมูกทุกอย่าง แต่ถ้ากำลังตรวจดูหน่วยรับความรู้สึก นี่เป็นระบบที่ดีที่สุดในโลก"[29][30][31]
ความหลากหลาย
แก้มีหน่วยรับกลิ่นหลายประเภทมาก โดยสัตว์เลี้ยงลูกด้วยนมอาจมียีนเป็นจำนวนถึง 1,000 ยีน ซึ่งเป็นร้อยละ 3 ทั้งหมดของจีโนม แต่ยีนหน่วยรับกลิ่นทั้งหมดก็ไม่ได้แสดงออกและไม่ทำงาน ตามการวิเคราะห์ข้อมูลของโครงการจีโนมมนุษย์ มนุษย์มียีนหน่วยรับกลิ่นที่ทำงานได้ประมาณ 400 ยีน โดยยีนที่เหลืออีก 600 เป็นยีนเทียม (pseudogene)[32]
เหตุผลที่มีหน่วยรับกลิ่นหลายประเภทมากก็เพื่อสร้างระบบที่สามารถแยกแยะกลิ่นให้มากที่สุดเท่าที่จะเป็นไปได้ โดยหน่วยรับกลิ่นแต่ละประเภทก็ไม่ใช่แค่รับกลิ่นชนิดเดียว เพราะสามารถตรวจจับโครงสร้างกลิ่นที่คล้ายกันเป็นจำนวนหนึ่งได้[33][34]
คล้ายกับระบบภูมิคุ้มกัน ความหลายหลายของประเภทหน่วยรับกลิ่นจะช่วยให้สามารถกำหนดกลิ่นที่ไม่เคยได้มาก่อน แต่ระบบภูมิคุ้มกันก็ยังสร้างความหลากหลายผ่านกระบวนการ V(D)J recombination ซึ่งไม่เหมือนกับหน่วยรับกลิ่นที่แปรรหัสมาจากยีนหนึ่ง ๆ โดยเฉพาะ ดังนั้น จึงมีจีโนมค่อนข้างหใญ่ที่อุทิศเพื่อเข้ารหัสยีน OR นอกจากนั้น กลิ่นโดยมากจะทำให้หน่วยรับกลิ่นมากกว่าหนึ่งชนิดเริ่มทำงาน เนื่องจากการรวมผสมกัน (combination) และการเรียงสับเปลี่ยน (permutation) ของหน่วยรับกลิ่นมีจำนวนมาก ดังนั้น ระบบรับกลิ่นจึงสามารถตรวจจับแยกแยะโมเลกุลกลิ่นเป็นจำนวนมาก
การกำหนดลิแกนด์ (Deorphanization) ของหน่วยรับกลิ่นสามารถทำได้ผ่านเทคนิคทางสรีรวิทยาไฟฟ้าและการสร้างภาพ เพื่อวิเคราะห์รูปแบบการตอบสนองของเซลล์ประสาทรับกลิ่นตัวเดียวต่อกลิ่นต่าง ๆ[35]
ข้อมูลเช่นนี้จะทำให้สามารถถอดรหัสเชิงผสม (combinational code) ของการได้กลิ่น[36]
ความหลากหลายทางการแสดงออกของ OR เยี่ยงนี้จะเพิ่มสมรรถภาพการได้กลิ่นให้มากที่สุด ทั้งการแสดงออกเป็นอัลลีลเดียว (monoallelic) ของ OR ในเซลล์ประสาทหนึ่ง ๆ และการแสดงออกของ OR หลายประเภทมากที่สุดในกลุ่มนิวรอน จะสำคัญต่อทั้งความจำเพาะและความไวในการได้กลิ่น ดังนั้น การทำงานของหน่วยรับกลิ่นจึงเป็นการแก้ปัญหาสองอย่าง
งานศึกษาที่ใช้การจำลองทางคณิตศาสตร์และการจำลองโดยคอมพิวเตอร์ได้เสนอกลไกควบคุม 3 ชั้นที่ดีสุดตามวิวัฒนาการ รวมทั้ง zonal segregation, epigenetic barrier crossing coupled to a negative feedback loop, และ enhancer competition step[37] แบบจำลองนี้ไม่เพียงบ่งชี้ให้มีการแสดงออกของ OR แบบอัลลีลเดียวในเซลล์เดียว แต่ยังชี้แจงด้วยว่า ระบบรับกลิ่นจะสร้างและรักษาความหลายหลายสูงสุดของการแสดงออกของยีน OR ได้อย่างไร
หมู่ (family)
แก้มีการพัฒนาตั้งชื่อยีนสำหรับหมู่หน่วยรับกลิ่น[38] ซึ่งกลายเป็นจุดเริ่มต้นของสัญลักษณ์ยีนที่เข้ารหัสหน่วยรับกลิ่นในโครงการจีโนมมนุษย์ คือ ชื่อของสมาชิกหน่วยรับกลิ่นแต่ละตัวในหมู่จะอยู่ในรูปแบบ "ORnXm" ที่
- OR เป็นรากของชื่อ (Olfactory Receptor superfamily)
- n = จำนวนเต็มที่ระบุหมู่ (family เช่น 1-56) ซึ่งสมาชิกมีลำดับโปรตีนเหมือนกันมากกว่า 40%
- X = อักษรเดี่ยว (A, B, C, ...) ที่กำหนดหมู่ย่อย (subfamily) ที่สมาชิกมีลำดับโปรตีนเหมือนกันมากกว่า 60%
- m = จำนวนเต็มที่กำหนดไอโซฟอร์ม (isoform) ของโปรตีนที่เป็นสมาชิกแต่ละตัวในหมู่
ยกตัวอย่างเช่น OR1A1 เป็นไอโซฟอร์มแรกของหมู่ย่อย A ของหน่วยรับกลิ่นกลุ่ม 1 สมาชิกที่อยู่ในหมู่ย่อยเดียวกัน (คือมีลำดับโปรตีน >60% เหมือนกัน) น่าจะรู้โมเลกุลกลิ่นที่มีโครงสร้างคล้าย ๆ กัน[39]
มีการแบ่งหน่วยรับกลิ่นในมนุษย์ออกเป็น 2 ชั้น (class) คือ[40]
- class I (หน่วยรับกลิ่นคล้ายของปลา) OR families 51-56
- class II (หน่วยรับกลิ่นเฉพาะต่อสัตว์สี่ขา) OR families 1-13
วิวัฒนาการ
แก้หน่วยรับกลิ่นเป็นหมู่ยีน (multigene) ที่มีลำดับเหมือนกันเป็นบางส่วน (evolutionarily conserved) ข้ามสัตว์มีกระดูกสันหลังสปีชีส์ต่าง ๆ[4] แม้ยีนที่แสดงออกเป็นหน่วยรับกลิ่นที่ใช้ได้จะมีจำนวนต่าง ๆ กัน โดยหนูหริ่งมียีน OR ที่เข้ารหัสโปรตีนถึง 1,035 ยีน เทียบกับมนุษย์ที่มีเพียงแค่ 387 ยีน[6] หน่วยรับกลิ่นเป็นสมาชิกของ G protein-coupled receptor แม้จะมีลำดับต่างหาก ๆ ที่ไม่เหมือนกับสมาชิกกลุ่มอื่น ๆ[4]
หมู่ยีนหน่วยรับกลิ่นของสัตว์มีกระดูกสันหลังพบว่า ได้วิวัฒนาการผ่านเหตุการณ์ทางจีโนมรวมทั้งการเพิ่มยีน (gene duplication) และการเปลี่ยนยีน (gene conversion)[41] หลักฐานของการเพิ่มยีนแบบเรียงตามกันเป็นคู่ (tandem duplication) มาจากความจริงว่า ยีนหน่วยรับกลิ่นเป็นจำนวนมาก่ในสัตว์เคลดเดียวกัน ก็จะอยู่ในกลุ่มยีน (gene cluster) เดียวกัน[42] จนถึงทุกวันนี้ การจัดระเบียบกลุ่มยีนของ OR ก็ยังเหมือนกันมากระหว่างมนุษย์กับหนูหริ่ง แม้จำนวน OR ที่ทำงานได้จริง ๆ จะต่างกันมากระหว่างสองสปีชีส์นี้[43] วิวัฒนาการแบบเกิดตาย (birth-and-death evolution) เช่นนี้ได้รวมส่วนต่าง ๆ จากยีน OR หลายยีน เป็นการสร้างและทำลายรูปแบบต่าง ๆ ของจุดยึดกลิ่น (odorant binding site) เป็นการสร้างยีน OR ที่ทำงานได้ใหม่ ๆ พร้อมกับยีนเทียม[44]
เทียบกับสัตว์เลี้ยงลูกด้วยนมอื่น ๆ ไพรเมตมียีน OR ที่ทำงานได้ค่อนข้างน้อย ยกตัวอย่างเช่น ตั้งแต่แยกออกจากบรรพบุรุษเดียวกันล่าสุด (MRCA) หนูหริ่งได้เพิ่มยีน OR 623 ยีน และสูญยีน 285 ยีน เทียบกับมนุษย์ที่ได้ยีนเพิ่มเพียง 83 ยีน แต่สูญถึง 428 ยีน[6] หนูหริ่งมียีน OR ที่เข้ารหัสโปรตีน 1,035 ยีน เทียบกับมนุษย์ที่มีเพียงแค่ 387 ยีน[6] สมมติฐานความสำคัญของการเห็น (vision priority hypothesis) เสนอว่า วิวัฒนาการของการเห็นเป็นสีในไพรเมตอาจลดการพึ่งพาอาศัยการได้กลิ่น ซึ่งอธิบายความกดดันทางการคัดเลือกโดยธรรมชาติที่ลดลง และอธิบายการสะสมเพิ่มยีนเทียมของหน่วยรับกลิ่นในไพรเมต[45]
แต่งานศึกษาปี 2006 ได้หักล้างสมมติฐานนี้โดยแสดงว่า เป็นสมมติฐานที่อาศัยข้อมูลและข้อสมมุติที่ทำให้เขว เพราะสมมติฐานสมมุติว่า ยีน OR ที่ทำงานได้จริง ๆ สัมพันธ์กับสมรรถภาพของการได้กลิ่นของสัตว์[45] ในมุมมองนี้ การลดอัตราส่วนยีน OR ที่ทำงานได้จะลดการได้กลิ่น ดังนั้น สปีชีส์ที่มีจำนวนยีนเทียมสูงกว่าก็จะมีสมรรถภาพการได้กลิ่นน้อยกว่า แต่ข้อสมมุตินี้ผิดพลาด เพราะสุนัขที่ชื่อว่ามีจมูกดี[46] กลับไม่ได้มียีน OR ที่ทำงานได้จำนวนมากที่สุด[6] (ประมาณ 1,000 ยีน) นอกจากนั้นยีนเทียมยังอาจทำหน้าที่บางอย่างได้ ยีนเทียม 67% ของ OR ในมนุษย์จะแสดงออกที่เยื่อรับกลิ่น ซึ่งอาจมีหน้าที่ควบคุมกระบวนการแสดงออกของยีน[47]
ยิ่งกว่านั้น สมมติฐานความสำคัญของการเห็นสมมุติการเสียยีน OR ที่ทำงานได้อย่างสำคัญเมื่อลิงโลกเก่าแยกสายพันธุ์ออก แต่ข้อสรุปนี้มีความเอนเอียงเนื่องกับข้อมูลที่มีรายละเอียดน้อยจากยีน OR เพียงแค่ 100 ยีน[48] และงานศึกษาที่มีรายละเอียดสูงก็สนับสนุนว่า ไพรเมตได้เสียยีน OR ในทุก ๆ สายพันธุ์ทั้งลิงโลกเก่าและลิงโลกใหม่ เริ่มตั้งแต่บรรพบุรุษล่าสุดร่วมกันจนถึงมนุษย์ ซึ่งบ่งว่า การเสียยีน OR ในไพรเมตไม่สามารถอธิบายได้ทั้งหมดด้วยการเปลี่ยนสมรรถภาพของการเห็น[49] เพราะลิงโลกเก่าเห็นเป็นสีดีกว่าลิงโลกใหม่โดยมาก
นักวิชาการยังแสดงด้วยว่า การไร้ความกดดันทางการคัดเลือกต่อหน่วยรับกลิ่นก็ยังมีอยู่ในมนุษย์ปัจจุบัน ซึ่งแสดงนัยว่า ยังไม่ถึงจุดการทำงานในระดับต่ำสุดดั่งเป็นที่ราบ และดังนั้น สมรรถภาพการได้กลิ่นก็ยังอาจจะลดลงเรื่อย ๆ นี่พิจารณาว่า เป็นเบาะแสเบื้องต้นที่แสดงทิศทางวิวัฒนาการในอนาคตของมนุษย์[50]
การค้นพบและประวัติ
แก้ในปี 2004 ศ. ดร. ลินดา บี บัก และ ศ. ดร. ริชาร์ด แอกเซิล ได้รับรางวัลโนเบลสาขาสรีรวิทยาหรือการแพทย์[51] เพราะได้ค้นพบมูลฐานทางพันธุกรรมของหน่วยรับกลิ่น[52]
ในปี 2006 นักวิชาการได้พบว่า ยังมีชั้นหน่วยรับกลิ่นอีกชั้นหนึ่ง ซึ่งเรียกว่า trace amine-associated receptors (TAARs) ที่ใช้ตรวจจับ amine ที่ระเหยได้[53] แม้จะเป็น G protein-coupled receptor เหมือนกัน แต่ลำดับโปรตีนก็ไม่สัมพันธ์กับหน่วยรับกลิ่นดังที่กล่าวมาแล้ว เป็นโปรตีนที่เข้ารหัสโดยยีนกลุ่มเล็ก ๆ ที่พบในมนุษย์ หนูหริ่ง และปลา งานศึกษาในหนูหริ่งพบหน่วยรับกลิ่นเช่นนี้ 14 ชนิด ซึ่งสามารถตรวจจับ amine ที่ระเหยได้ซึ่งพบอย่างเข้มข้นในฉี่ของหนูตัวผู้ ซึ่งแสดงนัยว่า นี่อาจเป็นหน่วยรับกลิ่นชั้นที่ทำหน้าที่ต่างหาก โดยอาจจะเนื่องกับกลิ่นทางสังคม[4]
การแสดงออกของหน่วยรับกลิ่นที่ใช้งานได้โดยเทคนิค heterologous expression ที่จำกัดในสปีชีส์อื่น ๆ ทำให้วิเคราะห์การตอบสนองของเซลล์รับกลิ่นเดี่ยว ๆ ต่อกลิ่นต่าง ๆ ได้ยาก หน่วยรับกลิ่นที่สร้างผ่านพันธุวิศกรรม คือ OR-I7 เป็นตัวแรกที่ได้ตรวจสอบ เพื่อระบุขอบเขตการตอบสนองต่อกลิ่นของตัวรับแอลดีไฮด์ที่มีอยู่ตามธรรมชาติ[35]
ดูเพิ่ม
แก้เชิงอรรถและอ้างอิง
แก้- ↑ Bjarnadottir, TIC; Gloriam, DE; Hellstrand, SH; Kristiansson, H; Fredriksson, R; Schioth, HB (September 2006). "Comprehensive repertoire and phylogenetic analysis of the G protein- coupled receptors in human and mouse". Genomics. 88 (3): 263–73. doi:10.1016/j.ygeno.2006.04.001. PMID 16753280.
{{cite journal}}: CS1 maint: uses authors parameter (ลิงก์) - ↑ Gaillard I, Rouquier S, Giorgi D (February 2004). "Olfactory receptors". Cellular and Molecular Life Sciences. 61 (4): 456–69. doi:10.1007/s00018-003-3273-7. PMID 14999405. S2CID 18608331.
- ↑ Hussain A, Saraiva LR, Korsching SI (March 2009). "Positive Darwinian selection and the birth of an olfactory receptor clade in teleosts". Proceedings of the National Academy of Sciences of the United States of America. 106 (11): 4313–8. Bibcode:2009PNAS..106.4313H. doi:10.1073/pnas.0803229106. PMC 2657432. PMID 19237578.
- ↑ 4.0 4.1 4.2 4.3 4.4 4.5 Buck & Bargmann 2013, Mammals Share a Large Family of Odorant Receptors, 714-715
- ↑ Niimura Y (December 2009). "Evolutionary dynamics of olfactory receptor genes in chordates: interaction between environments and genomic contents". Human Genomics. 4 (2): 107–18. doi:10.1186/1479-7364-4-2-107. PMC 3525206. PMID 20038498.
- ↑ 6.0 6.1 6.2 6.3 6.4 Niimura Y, Nei M (August 2007). "Extensive gains and losses of olfactory receptor genes in mammalian evolution". PLOS ONE. 2 (8): e708. Bibcode:2007PLoSO...2..708N. doi:10.1371/journal.pone.0000708. PMC 1933591. PMID 17684554.
- ↑ Rinaldi A (July 2007). "The scent of life. The exquisite complexity of the sense of smell in animals and humans". EMBO Reports. 8 (7): 629–33. doi:10.1038/sj.embor.7401029. PMC 1905909. PMID 17603536.
- ↑ Buck & Bargmann 2013, Different Combinations of Receptors Encode Different Odorants, 715-716
- ↑ Gu X, Karp PH, Brody SL, Pierce RA, Welsh MJ, Holtzman MJ, Ben-Shahar Y (March 2014). "Chemosensory functions for pulmonary neuroendocrine cells". American Journal of Respiratory Cell and Molecular Biology. 50 (3): 637–46. doi:10.1165/rcmb.2013-0199OC. PMC 4068934. PMID 24134460.
- ↑ Hallem EA, Dahanukar A, Carlson JR (2006). "Insect odor and taste receptors". Annual Review of Entomology. 51: 113–35. doi:10.1146/annurev.ento.51.051705.113646. PMID 16332206.
- ↑ Spehr M, Schwane K, Riffell JA, Zimmer RK, Hatt H (May 2006). "Odorant receptors and olfactory-like signaling mechanisms in mammalian sperm". Molecular and Cellular Endocrinology. 250 (1–2): 128–36. doi:10.1016/j.mce.2005.12.035. PMID 16413109. S2CID 45545572.
- ↑ Buck & Bargmann 2013, Odorants Are Encoded in the Nose by Dispersed Neurons, 716-717
- ↑ Khafizov K, Anselmi C, Menini A, Carloni P (March 2007). "Ligand specificity of odorant receptors". Journal of Molecular Modeling. 13 (3): 401–9. doi:10.1007/s00894-006-0160-9. PMID 17120078. S2CID 604107.
- ↑ de March CA, Kim SK, Antonczak S, Goddard WA, Golebiowski J (September 2015). "G protein-coupled odorant receptors: From sequence to structure". Protein Science. 24 (9): 1543–8. doi:10.1002/pro.2717. PMC 4570547. PMID 26044705.
- ↑ de March CA, Yu Y, Ni MJ, Adipietro KA, Matsunami H, Ma M, Golebiowski J (July 2015). "Conserved Residues Control Activation of Mammalian G Protein-Coupled Odorant Receptors". Journal of the American Chemical Society. 137 (26): 8611–6. doi:10.1021/jacs.5b04659. PMC 4497840. PMID 26090619.
- ↑ Wang J, Luthey-Schulten ZA, Suslick KS (March 2003). "Is the olfactory receptor a metalloprotein?". Proceedings of the National Academy of Sciences of the United States of America. 100 (6): 3035–9. Bibcode:2003PNAS..100.3035W. doi:10.1073/pnas.262792899. PMC 152240. PMID 12610211.
- ↑ Crabtree RH (1978). "Copper (I): A possible olfactory binding site". Journal of Inorganic and Nuclear Chemistry. 40 (7): 1453. doi:10.1016/0022-1902(78)80071-2.
- ↑ Duan X, Block E, Li Z, Connelly T, Zhang J, Huang Z, Su X, Pan Y, Wu L, Chi Q, Thomas S, Zhang S, Ma M, Matsunami H, Chen GQ, Zhuang H (February 2012). "Crucial role of copper in detection of metal-coordinating odorants". Proceedings of the National Academy of Sciences of the United States of America. 109 (9): 3492–7. Bibcode:2012PNAS..109.3492D. doi:10.1073/pnas.1111297109. PMC 3295281. PMID 22328155.
- ↑ Buck LB (November 2004). "Olfactory receptors and odor coding in mammals". Nutrition Reviews. 62 (11 Pt 2): S184–8, discussion S224–41. doi:10.1301/nr.2004.nov.S184-S188. PMID 15630933.
- ↑ Saberi M, Seyed-Allaei H (April 2016). "Odorant receptors of Drosophila are sensitive to the molecular volume of odorants". Scientific Reports. 6: 25103. Bibcode:2016NatSR...625103S. doi:10.1038/srep25103. PMC 4844992. PMID 27112241.
- ↑ Jones DT, Reed RR (May 1989). "Golf: an olfactory neuron specific-G protein involved in odorant signal transduction". Science. 244 (4906): 790–5. Bibcode:1989Sci...244..790J. doi:10.1126/science.2499043. PMID 2499043.
- ↑ 22.0 22.1 Purves et al 2008a, The Transduction of Olfactory Signals, pp. 375-378
- ↑ Brookes JC, Hartoutsiou F, Horsfield AP, Stoneham AM (January 2007). "Could humans recognize odor by phonon assisted tunneling?". Physical Review Letters. 98 (3): 038101. arXiv:physics/0611205. Bibcode:2007PhRvL..98c8101B. doi:10.1103/PhysRevLett.98.038101. PMID 17358733. S2CID 1519986.
- ↑ Franco MI, Turin L, Mershin A, Skoulakis EM (March 2011). "Molecular vibration-sensing component in Drosophila melanogaster olfaction". Proceedings of the National Academy of Sciences of the United States of America. 108 (9): 3797–802. Bibcode:2011PNAS..108.3797F. doi:10.1073/pnas.1012293108. PMC 3048096. PMID 21321219.
- ↑ Schramm VL (October 2007). "Binding isotope effects: boon and bane". Current Opinion in Chemical Biology. 11 (5): 529–36. doi:10.1016/j.cbpa.2007.07.013. PMC 2066183. PMID 17869163.
- ↑ Gane S, Georganakis D, Maniati K, Vamvakias M, Ragoussis N, Skoulakis EM, Turin L (2013). "Molecular vibration-sensing component in human olfaction". PLOS ONE. 8 (1): e55780. Bibcode:2013PLoSO...855780G. doi:10.1371/journal.pone.0055780. PMC 3555824. PMID 23372854.
- ↑ Block E, Jang S, Matsunami H, Sekharan S, Dethier B, Ertem MZ, Gundala S, Pan Y, Li S, Li Z, Lodge SN, Ozbil M, Jiang H, Penalba SF, Batista VS, Zhuang H (May 2015). "Implausibility of the vibrational theory of olfaction". Proceedings of the National Academy of Sciences of the United States of America. 112 (21): E2766-74. Bibcode:2015PNAS..112E2766B. doi:10.1073/pnas.1503054112. PMC 4450420. PMID 25901328.
- ↑ Vosshall LB (May 2015). "Laying a controversial smell theory to rest". Proceedings of the National Academy of Sciences of the United States of America. 112 (21): 6525–6. Bibcode:2015PNAS..112.6525V. doi:10.1073/pnas.1507103112. PMC 4450429. PMID 26015552.
- ↑ Everts S (2015). "Receptor Research Reignites A Smelly Debate". Chemical & Engineering News. 93 (18): 29–30.
- ↑ Turin L, Gane S, Georganakis D, Maniati K, Skoulakis EM (June 2015). "Plausibility of the vibrational theory of olfaction". Proceedings of the National Academy of Sciences of the United States of America. 112 (25): E3154. Bibcode:2015PNAS..112E3154T. doi:10.1073/pnas.1508035112. PMC 4485082. PMID 26045494.
- ↑ Block E, Jang S, Matsunami H, Batista VS, Zhuang H (June 2015). "Reply to Turin et al.: Vibrational theory of olfaction is implausible". Proceedings of the National Academy of Sciences of the United States of America. 112 (25): E3155. Bibcode:2015PNAS..112E3155B. doi:10.1073/pnas.1508443112. PMC 4485112. PMID 26045493.
- ↑ Gilad Y, Lancet D (March 2003). "Population differences in the human functional olfactory repertoire". Molecular Biology and Evolution. 20 (3): 307–14. doi:10.1093/molbev/msg013. PMID 12644552.
- ↑ Malnic B, Hirono J, Sato T, Buck LB (March 1999). "Combinatorial receptor codes for odors". Cell. 96 (5): 713–23. doi:10.1016/S0092-8674(00)80581-4. PMID 10089886. S2CID 12335310.
- ↑ Araneda RC, Peterlin Z, Zhang X, Chesler A, Firestein S (March 2004). "A pharmacological profile of the aldehyde receptor repertoire in rat olfactory epithelium". The Journal of Physiology. 555 (Pt 3): 743–56. doi:10.1113/jphysiol.2003.058040. PMC 1664868. PMID 14724183.
- ↑ 35.0 35.1 Smith R, Peterlin Z, Araneda R (2013). Pharmacology of Mammalian Olfactory Receptors. Methods in Molecular Biology. Vol. 1003. Olfactory Receptors Methods in Molecular Biology: Humana Press. pp. 203–209. doi:10.1007/978-1-62703-377-0_15. ISBN 978-1-62703-377-0. PMC 8529646. PMID 23585044.
- ↑ de March CA, Ryu S, Sicard G, Moon C, Golebiowski J (September 2015). "Structure–odour relationships reviewed in the postgenomic era". Flavour and Fragrance Journal. 30 (5): 342–361. doi:10.1002/ffj.3249.
- ↑ Tian XJ, Zhang H, Sannerud J, Xing J (May 2016). "Achieving diverse and monoallelic olfactory receptor selection through dual-objective optimization design". Proceedings of the National Academy of Sciences of the United States of America (ภาษาอังกฤษ). 113 (21): E2889-98. arXiv:1505.05179. Bibcode:2016PNAS..113E2889T. doi:10.1073/pnas.1601722113. PMC 4889386. PMID 27162367.
- ↑ Glusman G, Bahar A, Sharon D, Pilpel Y, White J, Lancet D (November 2000). "The olfactory receptor gene superfamily: data mining, classification, and nomenclature". Mammalian Genome. 11 (11): 1016–23. CiteSeerX 10.1.1.592.3303. doi:10.1007/s003350010196. PMID 11063259. S2CID 7573615.
- ↑ Malnic B, Godfrey PA, Buck LB (February 2004). "The human olfactory receptor gene family". Proceedings of the National Academy of Sciences of the United States of America. 101 (8): 2584–9. Bibcode:2004PNAS..101.2584M. doi:10.1073/pnas.0307882100. PMC 356993. PMID 14983052.
- ↑ Freeitag J, Krieger J, Strotmann J, Breer H (1995). "Two classes of olfactory receptors in Canopus laevis". Neuron. 15 (6): 1383–1392. doi:10.1016/0896-6273(95)90016-0. PMID 8845161.
- ↑ Nei M, Rooney AP (2005). "Concerted and birth-and-death evolution of multigene families". Annual Review of Genetics. 39: 121–52. doi:10.1146/annurev.genet.39.073003.112240. PMC 1464479. PMID 16285855.
- ↑ Niimura Y, Nei M (2006). "Evolutionary dynamics of olfactory and other chemosensory receptor genes in vertebrates". Journal of Human Genetics. 51 (6): 505–17. doi:10.1007/s10038-006-0391-8. PMC 1850483. PMID 16607462.
- ↑ Niimura Y, Nei M (February 2005). "Comparative evolutionary analysis of olfactory receptor gene clusters between humans and mice". Gene. 346 (6): 13–21. doi:10.1016/j.gene.2004.09.025. PMID 15716120.
- ↑ Nozawa M, Nei M (2008). "Genomic drift and copy number variation of chemosensory receptor genes in humans and mice". Cytogenetic and Genome Research. 123 (1–4): 263–9. doi:10.1159/000184716. PMC 2920191. PMID 19287163.
- ↑ 45.0 45.1 Gilad Y, Wiebe V, Przeworski M, Lancet D, Pääbo S (January 2004). "Loss of olfactory receptor genes coincides with the acquisition of full trichromatic vision in primates". PLOS Biology. 2 (1): E5. doi:10.1371/journal.pbio.0020005. PMC 314465. PMID 14737185.
- ↑ Craven BA, Paterson EG, Settles GS (June 2010). "The fluid dynamics of canine olfaction: unique nasal airflow patterns as an explanation of macrosmia". Journal of the Royal Society, Interface. 7 (47): 933–43. doi:10.1098/Rsif.2009.0490. PMC 2871809. PMID 20007171.
- ↑ Zhang X, De la Cruz O, Pinto JM, Nicolae D, Firestein S, Gilad Y (2007). "Characterizing the expression of the human olfactory receptor gene family using a novel DNA microarray". Genome Biology. 8 (5): R86. doi:10.1186/gb-2007-8-5-r86. PMC 1929152. PMID 17509148.
- ↑ Matsui A, Go Y, Niimura Y (May 2010). "Degeneration of olfactory receptor gene repertories in primates: no direct link to full trichromatic vision". Molecular Biology and Evolution. 27 (5): 1192–200. doi:10.1093/molbev/msq003. PMID 20061342.
- ↑ Niimura Y (April 2012). "Olfactory receptor multigene family in vertebrates: from the viewpoint of evolutionary genomics". Current Genomics. 13 (2): 103–14. doi:10.2174/138920212799860706. PMC 3308321. PMID 23024602.
- ↑ Pierron D, Cortés NG, Letellier T, Grossman LI (February 2013). "Current relaxation of selection on the human genome: tolerance of deleterious mutations on olfactory receptors". Molecular Phylogenetics and Evolution. 66 (2): 558–64. doi:10.1016/j.ympev.2012.07.032. PMID 22906809.
- ↑ "Press Release: The 2004 Nobel Prize in Physiology or Medicine". สืบค้นเมื่อ 2007-06-06.
- ↑ Buck L, Axel R (April 1991). "A novel multigene family may encode odorant receptors: a molecular basis for odor recognition". Cell. 65 (1): 175–87. doi:10.1016/0092-8674(91)90418-X. PMID 1840504.
- ↑ Liberles SD, Buck LB (August 2006). "A second class of chemosensory receptors in the olfactory epithelium". Nature. 442 (7103): 645–50. Bibcode:2006Natur.442..645L. doi:10.1038/nature05066. PMID 16878137. S2CID 2864195.